L'équipe de recherche de l'Université de Glasgow a développé un nouveau modèle de langage protéique PLM-Interact, capable de prédire efficacement les interactions protéiques et d'analyser les impacts des mutations.
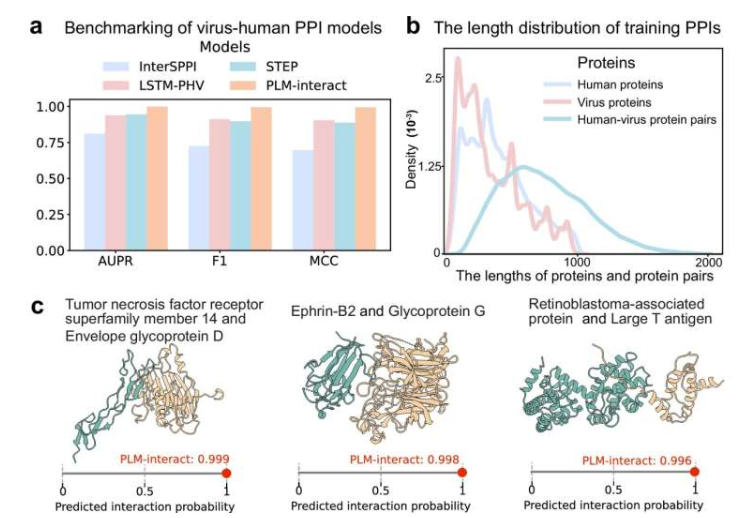
L'équipe de recherche, dirigée par le Dr Ke Yuan de la Faculté des sciences du cancer, le professeur Craig Macdonald de la Faculté d'informatique et le professeur David L Robertson du Centre de recherche sur les virus, a développé le modèle de langage protéique PLM-Interact basé sur de grands modèles de langage. Ce modèle, entraîné sur plus de 421 000 paires d'interactions protéiques humaines, démontre une performance de prédiction supérieure aux modèles existants.
Le Dr Ke Yuan a déclaré : « Le supercalculateur DiRAC, initialement utilisé pour étudier les lois de la nature, nous aide maintenant à établir un nouveau modèle pour explorer les interactions protéiques. Les collègues de la Faculté d'informatique ont fourni un soutien en modélisation linguistique, et les ressources de calcul de DiRAC nous ont permis de compléter ce travail plus efficacement. » Le modèle de langage protéique a impliqué plus de 650 millions de paramètres lors de l'entraînement, utilisant le cluster GPU des installations de calcul haute performance DiRAC au Royaume-Uni pour accélérer la construction du modèle.
Le modèle de langage protéique PLM-Interact améliore la précision de prédiction des interactions protéiques de 16 % à 28 % par rapport à d'autres modèles IA avancés. Le modèle a réussi à prédire les interactions protéiques liées à cinq fonctions biologiques clés, alors que d'autres outils n'en prédisaient qu'une. L'étude confirme également que le modèle de langage protéique peut identifier précisément l'impact des mutations sur les interactions protéiques, y compris les types de mutations causant des maladies génétiques et le cancer.